Oficina de análise de dados NIR em ambiente R
Partes 4—5 - LDA e PLS
Ricardo Perdiz (Luz da Floresta) | 2021/07/02
Sumário
Recapitulação do primeiro dia
Interface para análise de dados do caret
Análise discriminante linear (LDA)
Sumário
Recapitulação do primeiro dia
Interface para análise de dados do caret
Análise discriminante linear (LDA)
Análise de mínimos quadrados parciais (PLS)
Recapitulação
Manipulação de dados utilizando o dplyr
Preparação de dados pré-análise
PCA
Figura de Wickham, H. e Grolemund, G. (2016) http://r4ds.had.co.nz.
Importando dados para a oficina
Opção 1 - Dados iris
dados <- irisOpção 2 - Importação de dados próprios
library("data.table")dados <- fread("MEUSDADOS.csv")Opção 3 - Conjunto de dados nir_data1, que acompanha o pacote NIRtools2.
dados <- fread("https://raw.githubusercontent.com/ricoperdiz/NIRtools/master/inst/extdata/nir_data.csv")Transformação do data.frame em um tibble1
library("tibble")dados <- as_tibble(dados)dados## # A tibble: 150 × 5## Sepal.Length Sepal.Width Petal.Length Petal.Width Species## <dbl> <dbl> <dbl> <dbl> <fct> ## 1 5.1 3.5 1.4 0.2 setosa ## 2 4.9 3 1.4 0.2 setosa ## 3 4.7 3.2 1.3 0.2 setosa ## 4 4.6 3.1 1.5 0.2 setosa ## 5 5 3.6 1.4 0.2 setosa ## 6 5.4 3.9 1.7 0.4 setosa ## 7 4.6 3.4 1.4 0.3 setosa ## 8 5 3.4 1.5 0.2 setosa ## 9 4.4 2.9 1.4 0.2 setosa ## 10 4.9 3.1 1.5 0.1 setosa ## # … with 140 more rows- Este passo NÃO É NECESSÁRIO. Ele facilita apenas a visualização dos dados no console, e indica o tipo de cada variável em seu conjunto de dados. Saiba mais sobre um
tibbleem https://r4ds.had.co.nz/tibbles.html.
Atenção!!!
Nova variável de nome de espécies
Em casos em que a variável contendo o nome da espécie apresente espaços, pontos . e/ou sinais diferentes, é necessário limparmos essa variável caso desejemos reter os valores de probabilidade atribuídos a cada espécie ao utilizarmos o caret.
Em casos assim, podemos tomar o procedimento abaixo:
library("dplyr")dados$SP1 <- case_when( dados$SP1 == "P. aracouchini" ~ "arac", dados$SP1 == "P. calanense" ~ "cala") %>% as.factor(.)Pré-processamento dos dados
Divisão dos dados em treino e teste
library("rsample") # initial_splitdados_split <- initial_split(dados, strata = "Species")treino <- training(dados_split)teste <- testing(dados_split)Criação e preparo da receita
library("recipes")treino_receita <- recipes::recipe(Species ~ ., data = treino) %>% prep()treino_receita## Recipe## ## Inputs:## ## role #variables## outcome 1## predictor 4## ## Training data contained 111 data points and no missing data.PCA
I - PCA com todos os dados?
Primeiro, criamos uma receita indicando no argumento data o nome de nosso conjunto de dados.
Depois fazemos as devidas atualizações dos papéis das variáveis que não são nem preditoras nem resposta com a função update_role().
Em seguida, executamos a PCA com a função step_pca() para todas as variáveis NIR, utilizando a função auxiliar all_predictors() para, por fim, prepararmos a receita com a função prep().
Depois, é só plotar os dados espremendo a receita, o que vai gerar uma PCA com os dados completos.
I - PCA com todos os dados?
dados_completos_receita_pca_pre <- recipes::recipe(Species ~ ., data = dados) %>% step_normalize(all_predictors()) %>% step_pca(all_predictors()) %>% prep()dados_completos_receita_pca_pre## Recipe## ## Inputs:## ## role #variables## outcome 1## predictor 4## ## Training data contained 150 data points and no missing data.## ## Operations:## ## Centering and scaling for Sepal.Length, Sepal.Width, Petal.Length, ... [trained]## PCA extraction with Sepal.Length, Sepal.Width, Petal.Length, P... [trained]II - PCA com todos os dados?
pca_tabela <- tidy(dados_completos_receita_pca_pre, 2)pca_tabela## # A tibble: 16 × 4## terms value component id ## <chr> <dbl> <chr> <chr> ## 1 Sepal.Length 0.521 PC1 pca_EX7FS## 2 Sepal.Width -0.269 PC1 pca_EX7FS## 3 Petal.Length 0.580 PC1 pca_EX7FS## 4 Petal.Width 0.565 PC1 pca_EX7FS## 5 Sepal.Length -0.377 PC2 pca_EX7FS## 6 Sepal.Width -0.923 PC2 pca_EX7FS## 7 Petal.Length -0.0245 PC2 pca_EX7FS## 8 Petal.Width -0.0669 PC2 pca_EX7FS## 9 Sepal.Length 0.720 PC3 pca_EX7FS## 10 Sepal.Width -0.244 PC3 pca_EX7FS## 11 Petal.Length -0.142 PC3 pca_EX7FS## 12 Petal.Width -0.634 PC3 pca_EX7FS## 13 Sepal.Length 0.261 PC4 pca_EX7FS## 14 Sepal.Width -0.124 PC4 pca_EX7FS## 15 Petal.Length -0.801 PC4 pca_EX7FS## 16 Petal.Width 0.524 PC4 pca_EX7FSIII - PCA com todos os dados?
Variação das PC's
sdev_pca_normalizada <- dados_completos_receita_pca_pre$steps[[2]]$res$sdevporcent_variacao_normalizada <- sdev_pca_normalizada^2 / sum(sdev_pca_normalizada^2)pca_normalizada_variacao <- data.frame( pcs = unique(pca_tabela$component), variacao_porcentagem = porcent_variacao_normalizada*100, variacao_cumulativa = cumsum(porcent_variacao_normalizada*100) )pca_normalizada_variacao## pcs variacao_porcentagem variacao_cumulativa## 1 PC1 72.9624454 72.96245## 2 PC2 22.8507618 95.81321## 3 PC3 3.6689219 99.48213## 4 PC4 0.5178709 100.00000IV - PCA com todos os dados?
Plotando PCA com dados completos
library("ggplot2")pc1 <- filter(pca_normalizada_variacao, pcs == "PC1")pc2 <- filter(pca_normalizada_variacao, pcs == "PC2")juice(dados_completos_receita_pca_pre) %>% ggplot(aes(PC1, PC2)) + geom_point(aes(color = Species), alpha = 0.7, size = 2) + xlab(paste0("PC1 (", round(pc1$variacao_porcentagem, 2), "%)")) + ylab(paste0("PC2 (", round(pc2$variacao_porcentagem, 2), "%)"))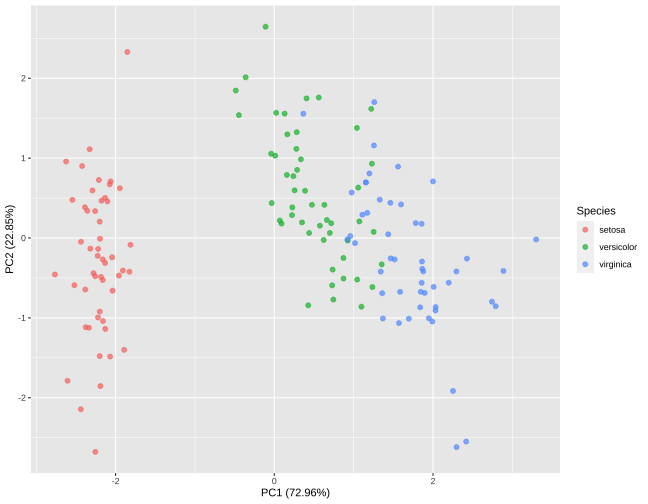
Pacote caret - Classification And REgression Training (Kuhn 2021)
Interface para análise de dados do caret
O pacote contem funções para simplificar tanto o treinamento de modelos para regressões e análises de classificação como a avaliação da efetividade dos modelos gerados.
Interface para análise de dados do caret
O pacote contem funções para simplificar tanto o treinamento de modelos para regressões e análises de classificação como a avaliação da efetividade dos modelos gerados.
Fluxo de processamento e análise de dados
Interface para análise de dados do caret
O pacote contem funções para simplificar tanto o treinamento de modelos para regressões e análises de classificação como a avaliação da efetividade dos modelos gerados.
Fluxo de processamento e análise de dados
Duas funções básicas:
caret::train()caret::trainControl()Análise discriminante linear (LDA)
Método de redução de dimensões ao mesmo tempo em que se retem o máximo de informação possível. O método ajuda a achar a combinação linear das variáveis originais que providenciam a melhor separação entre os grupos.1
Análise discriminante linear (LDA)
Método de redução de dimensões ao mesmo tempo em que se retem o máximo de informação possível. O método ajuda a achar a combinação linear das variáveis originais que providenciam a melhor separação entre os grupos.1
Maximiza a medida inter-classes e minimiza a medida intra-classes.
Análise discriminante linear (LDA)
Método de redução de dimensões ao mesmo tempo em que se retem o máximo de informação possível. O método ajuda a achar a combinação linear das variáveis originais que providenciam a melhor separação entre os grupos.1
Maximiza a medida inter-classes e minimiza a medida intra-classes.
Utilizada quando a variável dependente é categórica, e as variáveis independentes são quantitativas.
Análise discriminante linear (LDA)
Método de redução de dimensões ao mesmo tempo em que se retem o máximo de informação possível. O método ajuda a achar a combinação linear das variáveis originais que providenciam a melhor separação entre os grupos.1
Maximiza a medida inter-classes e minimiza a medida intra-classes.
Utilizada quando a variável dependente é categórica, e as variáveis independentes são quantitativas.
Em R, utilizamos a função lda() do pacote MASS (Venables e Ripley 2002)2.
Análise discriminante linear (LDA)
Método de redução de dimensões ao mesmo tempo em que se retem o máximo de informação possível. O método ajuda a achar a combinação linear das variáveis originais que providenciam a melhor separação entre os grupos.1
Maximiza a medida inter-classes e minimiza a medida intra-classes.
Utilizada quando a variável dependente é categórica, e as variáveis independentes são quantitativas.
Em R, utilizamos a função lda() do pacote MASS (Venables e Ripley 2002)2.
Usando o caret, a função é a mesma, porém dentro de uma interface comum a vários métodos diferentes3.
library("MASS")## ## Attaching package: 'MASS'## The following object is masked from 'package:dplyr':## ## selectFávero, L. P., Belfiore, P., Silva, F. L. e Chan, B.L. (2009) Análise de dados: Modelagem multivariada para tomada de decisões. Rio de Janeiro: Elsevier.
Venables, W. N. e Ripley, B. D. (2002) Modern Applied Statistics with S. Fourth Edition.
Lista completa de modelos aceitos no caret: https://topepo.github.io/caret/available-models.html.
Premissas1
- Normalidade multivariada
- Homogeneidade de variância/covariância
- Independência entre as observações
Premissas1
- Normalidade multivariada
- Homogeneidade de variância/covariância
- Independência entre as observações
Multicolinearidade pode diminuir o poder preditivo da classificação pois viola a independência das observações.
- Fávero, L. P., Belfiore, P., Silva, F. L. e Chan, B.L. (2009) Análise de dados: Modelagem multivariada para tomada de decisões. Rio de Janeiro: Elsevier.
Um exemplo - I - Execução
Receita criada no slide Criação da receita.
library("caret")ldafit_cvsimples <- train( treino_receita, treino, method = "lda", metric = "Accuracy" )Checagem de resultados
ldafit_cvsimples## Linear Discriminant Analysis ## ## 111 samples## 4 predictor## 3 classes: 'setosa', 'versicolor', 'virginica' ## ## Recipe steps: ## Resampling: Bootstrapped (25 reps) ## Summary of sample sizes: 111, 111, 111, 111, 111, 111, ... ## Resampling results:## ## Accuracy Kappa ## 0.9754789 0.9627761Um exemplo - II - Medindo performance do treino
getTrainPerf(ldafit_cvsimples)## TrainAccuracy TrainKappa method## 1 0.9754789 0.9627761 ldaUm exemplo - III - Predições e performance em teste
predicao_lda_basico <- ldafit_cvsimples %>% predict(., newdata = teste)predicao_lda_basico## [1] setosa setosa setosa setosa setosa setosa ## [7] setosa setosa setosa setosa setosa setosa ## [13] setosa versicolor versicolor versicolor virginica versicolor## [19] versicolor versicolor versicolor versicolor versicolor versicolor## [25] versicolor versicolor virginica virginica virginica virginica ## [31] virginica virginica virginica virginica versicolor virginica ## [37] virginica virginica virginica ## Levels: setosa versicolor virginicaUm exemplo - III - Predições e performance em teste
Para entender métricas de performance: https://topepo.github.io/caret/measuring-performance.html#class.
Um exemplo - IV - Matriz de confusão
Função confusao_lab() do pacote NIRtools1.
Caso não consigam ou não desejem instalar o pacote, basta fazer um source():
source("https://raw.githubusercontent.com/ricoperdiz/NIRtools/master/R/confusion_matrix.R")par(mar = c(1, 5, 5, 1))table(teste$Species, predicao_lda_basico) %>% confusao_lab(.)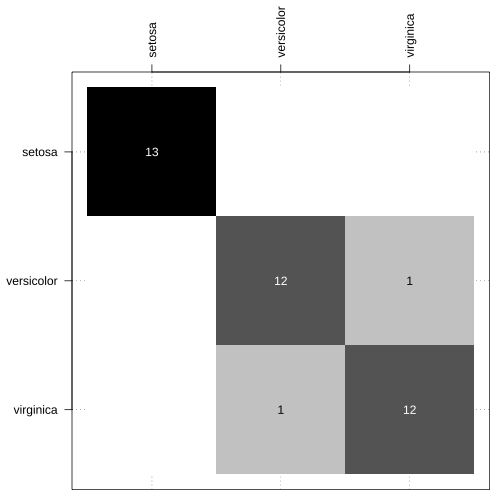
- Perdiz (2021) https://github.com/ricoperdiz/NIRtools
Argumentos1 para criação de modelos com a função trainControl()
method: boot, cv, LOOCV, LGOCV, repeatedcv e none (há mais opções!)numbererepeats: primeiro controla o número de divisões em cv/repeatedcv OU número de iterações para bootstrap e LOOCV; segundo se aplica somente ao k-fold.verboseIter: Mostra um logreturnData: Retorna os dados que servem de entrada?p: Porcentagem em LGOCVclassProbs: valor lógico para determinar se as probabilidades de atribuição a cada classe devem ser calculadasreturnResamp: all, final or noneallowParallel: Paraleliza?
- Para uma lista completa, acesse a ajuda da função:
?trainControl
Prática com trainControl()
Validação cruzada simples - cv
ctrl_cv <- trainControl(method = "cv", number = 10, # faz uma validacao 10-fold verboseIter = FALSE, returnData = TRUE, # Nao importante! Pode ser falso classProbs = TRUE, savePredictions = "all")ldafit_cv_10fold <- train( treino_receita, treino, method = "lda", trControl = ctrl_cv, metric = "Accuracy" # em classificacoes, dois valores sao possiveis: Accuracy ou Kappa )Acessando o resultado de train() - I
names(ldafit_cv_10fold)## [1] "method" "modelInfo" "modelType" "recipe" ## [5] "results" "pred" "bestTune" "call" ## [9] "dots" "metric" "control" "finalModel" ## [13] "trainingData" "resample" "resampledCM" "perfNames" ## [17] "maximize" "yLimits" "times" "levels" ## [21] "rs_seed"Para acessar as informações, basta executar um dos comandos abaixo:
ldafit_cv_10fold$method # metodo utilizado ldafit_cv_10fold$modelInfo # info do modeloldafit_cv_10fold$modelType # tipo de modeloldafit_cv_10fold$bestTuneldafit_cv_10fold$call # formula utilizada em train()ldafit_cv_10fold$dots # se verbose == TRUE ou FALSEldafit_cv_10fold$resample # estatisticas em cada reamostragemldafit_cv_10fold$metric # qual metrica utilizada em train()ldafit_cv_10fold$control # especificacoes de trainControl()Acessando o resultado de train() - II - Predições
ldafit_cv_10fold$pred %>% filter(obs != pred)## obs rowIndex pred setosa versicolor virginica## 1 versicolor 64 virginica 7.082366e-31 0.1304324 0.8695676## 2 virginica 99 versicolor 6.506261e-27 0.8282785 0.1717215## parameter Resample## 1 none Fold07## 2 none Fold10Acessando o resultado de train() - IV - resultados
ldafit_cv_10fold$results## parameter Accuracy Kappa AccuracySD KappaSD## 1 none 0.9818182 0.9723259 0.03833064 0.05834346Acessando o resultado de train() - V - Modelo final
Resultado da LDA, caso fizéssemos uso da função lda(), referente ao modelo final:
ldafit_cv_10fold$finalModel## Call:## lda(x, y)## ## Prior probabilities of groups:## setosa versicolor virginica ## 0.3333333 0.3333333 0.3333333 ## ## Group means:## Sepal.Length Sepal.Width Petal.Length Petal.Width## setosa 5.029730 3.440541 1.481081 0.2459459## versicolor 5.972973 2.751351 4.254054 1.3162162## virginica 6.610811 2.989189 5.616216 2.0405405## ## Coefficients of linear discriminants:## LD1 LD2## Sepal.Length 0.9107941 -0.6076288## Sepal.Width 1.3721594 2.7462192## Petal.Length -2.1944206 -0.2260993## Petal.Width -2.8023531 1.9159240## ## Proportion of trace:## LD1 LD2 ## 0.9893 0.0107Medindo performance do treino
getTrainPerf(ldafit_cv_10fold)## TrainAccuracy TrainKappa method## 1 0.9818182 0.9723259 ldaPredições e performance em teste
predicao_lda_cv_10fold <- ldafit_cv_10fold %>% predict(., newdata = teste)confusionMatrix(data = predicao_lda_cv_10fold, reference = teste$Species)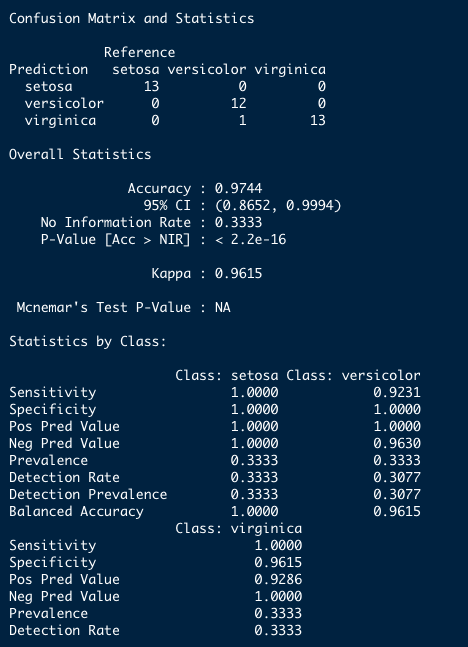
Matriz de confusão
Função confusao_lab() do pacote NIRtools1.
par(mar = c(1, 5, 5, 1))table(teste$Species, predicao_lda_basico) %>% NIRtools::confusao_lab(., add_CP = TRUE)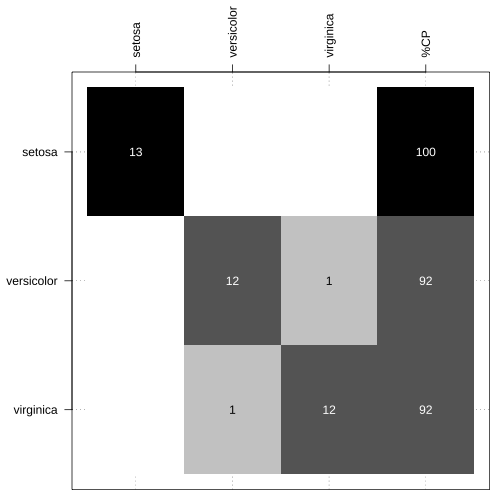
- Perdiz (2021) https://github.com/ricoperdiz/NIRtools
Validação cruzada repetida - repeatedcv
ctrl_lda_repeatedcv <- trainControl( method = "repeatedcv", number = 10, # faz uma validacao 10-fold repeats = 10, verboseIter = FALSE, returnData = FALSE, # Nao importante! Pode ser falso classProbs = TRUE, savePredictions = "all" )ldafit_repeatedcv_10fold_10vezes <- train( treino_receita, treino, method = "lda", trControl = ctrl_lda_repeatedcv, metric = "Accuracy" # em classificacoes, dois valores sao possiveis: Accuracy ou Kappa )Receitas de pré-processamento e o caret
Por que aprender isso?
Receitas de pré-processamento e o caret
Por que aprender isso?
Mais possibilidades no pré-processamento dos dados
Manter variáveis adicionais para medir performance
E o caret sem receita, pode?
Pode!
Execução
resultado_lda_sem_receita_basico <- train( Species ~ ., data = treino, method = "lda", metric = "Accuracy" )Checagem de resultado
print(resultado_lda_sem_receita_basico)## Linear Discriminant Analysis ## ## 111 samples## 4 predictor## 3 classes: 'setosa', 'versicolor', 'virginica' ## ## No pre-processing## Resampling: Bootstrapped (25 reps) ## Summary of sample sizes: 111, 111, 111, 111, 111, 111, ... ## Resampling results:## ## Accuracy Kappa ## 0.9667379 0.9495212Medindo performance do treino
getTrainPerf(resultado_lda_sem_receita_basico)## TrainAccuracy TrainKappa method## 1 0.9667379 0.9495212 ldaPredições e performance em teste
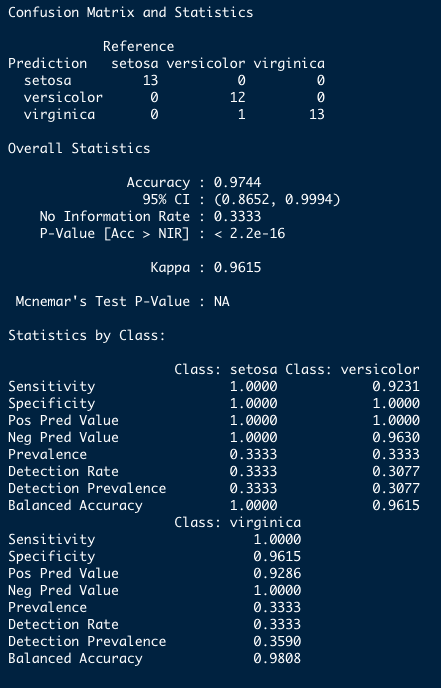
predicao_lda_sem_receita_basico <- resultado_lda_sem_receita_basico %>% predict(., newdata = teste)confusionMatrix(data = predicao_lda_sem_receita_basico, reference = teste$Species)## Confusion Matrix and Statistics## ## Reference## Prediction setosa versicolor virginica## setosa 13 0 0## versicolor 0 12 1## virginica 0 1 12## ## Overall Statistics## ## Accuracy : 0.9487 ## 95% CI : (0.8268, 0.9937)## No Information Rate : 0.3333 ## P-Value [Acc > NIR] : 7.509e-16 ## ## Kappa : 0.9231 ## ## Mcnemar's Test P-Value : NA ## ## Statistics by Class:## ## Class: setosa Class: versicolor Class: virginica## Sensitivity 1.0000 0.9231 0.9231## Specificity 1.0000 0.9615 0.9615## Pos Pred Value 1.0000 0.9231 0.9231## Neg Pred Value 1.0000 0.9615 0.9615## Prevalence 0.3333 0.3333 0.3333## Detection Rate 0.3333 0.3077 0.3077## Detection Prevalence 0.3333 0.3333 0.3333## Balanced Accuracy 1.0000 0.9423 0.9423Matriz de confusão
Função confusao_lab() do pacote NIRtools1.
par(mar = c(1, 5, 5, 1))table(teste$Species, predicao_lda_sem_receita_basico) %>% NIRtools::confusao_lab(.)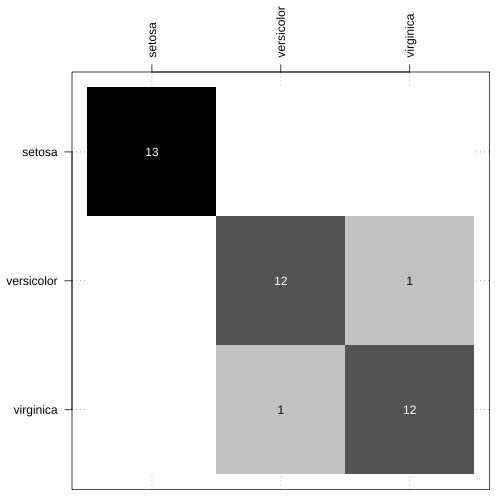
- Perdiz (2021) https://github.com/ricoperdiz/NIRtools
Análise de mínimos quadrados parciais (PLS)
Método muito utilizado na quimiometria, especialmente quando o número de variáveis preditoras é significantemente maior que o número de observações.
Análise de mínimos quadrados parciais (PLS)
Método muito utilizado na quimiometria, especialmente quando o número de variáveis preditoras é significantemente maior que o número de observações.
Baseia-se em variáveis latentes, em que cada fator é definido como uma combinação linear das variáveis originais das variáveis preditoras ou variável resposta.
Análise de mínimos quadrados parciais (PLS)
Método muito utilizado na quimiometria, especialmente quando o número de variáveis preditoras é significantemente maior que o número de observações.
Baseia-se em variáveis latentes, em que cada fator é definido como uma combinação linear das variáveis originais das variáveis preditoras ou variável resposta.
Essencialmente, trata-se de uma versão supervisionada da PCA1
library("pls")PLS com o pacote caret
Dados
Mesmo conjunto de dados utilizado anteriormente. Coloco aqui para facilitar a preparação dos dados para a PLS.
dados <- irisdados <- as_tibble(dados)dados_split <- initial_split(dados, strata = "Species")treino <- training(dados_split)teste <- testing(dados_split)treino_receita <- recipes::recipe(Species ~ ., data = treino) %>% prep()Execução
ctrl_exemplo_pls <- trainControl( method = "repeatedcv", number = 10, repeats = 10, verboseIter = FALSE, savePredictions = "final")plsfit_exemplo <- train( treino_receita, treino, method = "pls", metric = "Accuracy", trControl = ctrl_exemplo_pls, tuneLength = 3 )É importante comentar sobre a utilidade do argumento tuneGrid da função train() quando executamos uma PLS.
Também comentar sobre a possibilidade de paralelização da análise por meio do pacote paralell e a função registerDoParallel.
Checando o resultado
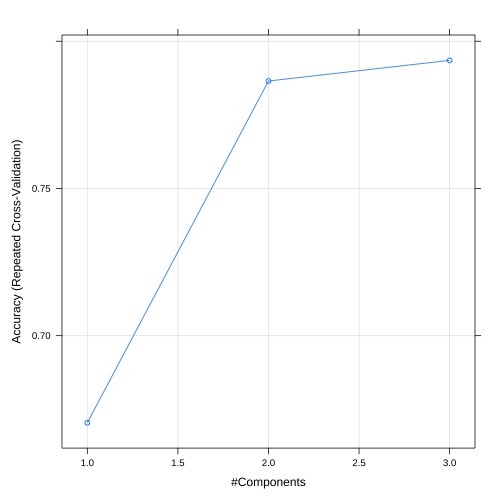
plsfit_exemplo## Partial Least Squares ## ## 111 samples## 4 predictor## 3 classes: 'setosa', 'versicolor', 'virginica' ## ## Recipe steps: ## Resampling: Cross-Validated (10 fold, repeated 10 times) ## Summary of sample sizes: 99, 99, 99, 101, 100, 99, ... ## Resampling results across tuning parameters:## ## ncomp Accuracy Kappa ## 1 0.6704242 0.5055691## 2 0.7864798 0.6787244## 3 0.7934949 0.6889434## ## Accuracy was used to select the optimal model using the largest value.## The final value used for the model was ncomp = 3.Predições e performance
plsfit_exemplo %>% predict(newdata = teste) %>% confusionMatrix(data = ., reference = teste$Species)## Confusion Matrix and Statistics## ## Reference## Prediction setosa versicolor virginica## setosa 13 0 0## versicolor 0 6 1## virginica 0 7 12## ## Overall Statistics## ## Accuracy : 0.7949 ## 95% CI : (0.6354, 0.907)## No Information Rate : 0.3333 ## P-Value [Acc > NIR] : 4.429e-09 ## ## Kappa : 0.6923 ## ## Mcnemar's Test P-Value : NA ## ## Statistics by Class:## ## Class: setosa Class: versicolor Class: virginica## Sensitivity 1.0000 0.4615 0.9231## Specificity 1.0000 0.9615 0.7308## Pos Pred Value 1.0000 0.8571 0.6316## Neg Pred Value 1.0000 0.7813 0.9500## Prevalence 0.3333 0.3333 0.3333## Detection Rate 0.3333 0.1538 0.3077## Detection Prevalence 0.3333 0.1795 0.4872## Balanced Accuracy 1.0000 0.7115 0.8269Validação cruzada Leave-one-out - LOOCV
ctrl_loocv <- trainControl( method = "LOOCV", verboseIter = FALSE, savePredictions = "final")plsfit_exemplo_loocv <- train( treino_receita, treino, method = "pls", metric = "Accuracy", trControl = ctrl_loocv, tuneGrid = expand.grid(ncomp = 2) )Checagem de resultado
plsfit_exemplo_loocv## Partial Least Squares ## ## 111 samples## 4 predictor## 3 classes: 'setosa', 'versicolor', 'virginica' ## ## Recipe steps: ## Resampling: Leave-One-Out Cross-Validation ## Summary of sample sizes: 110, 110, 110, 110, 110, 110, ... ## Resampling results:## ## Accuracy Kappa ## 0.7747748 0.6621622## ## Tuning parameter 'ncomp' was held constant at a value of 2Medindo performance do treino
getTrainPerf(plsfit_exemplo_loocv)## TrainAccuracy TrainKappa method## 1 0.7747748 0.6621622 plsPredições e performance em teste
predicao_plsfit_exemplo_loocv <- plsfit_exemplo_loocv %>% predict(., newdata = teste)confusionMatrix(data = predicao_plsfit_exemplo_loocv, reference = teste$Species)## Confusion Matrix and Statistics## ## Reference## Prediction setosa versicolor virginica## setosa 13 0 0## versicolor 0 6 2## virginica 0 7 11## ## Overall Statistics## ## Accuracy : 0.7692 ## 95% CI : (0.6067, 0.8887)## No Information Rate : 0.3333 ## P-Value [Acc > NIR] : 3.12e-08 ## ## Kappa : 0.6538 ## ## Mcnemar's Test P-Value : NA ## ## Statistics by Class:## ## Class: setosa Class: versicolor Class: virginica## Sensitivity 1.0000 0.4615 0.8462## Specificity 1.0000 0.9231 0.7308## Pos Pred Value 1.0000 0.7500 0.6111## Neg Pred Value 1.0000 0.7742 0.9048## Prevalence 0.3333 0.3333 0.3333## Detection Rate 0.3333 0.1538 0.2821## Detection Prevalence 0.3333 0.2051 0.4615## Balanced Accuracy 1.0000 0.6923 0.7885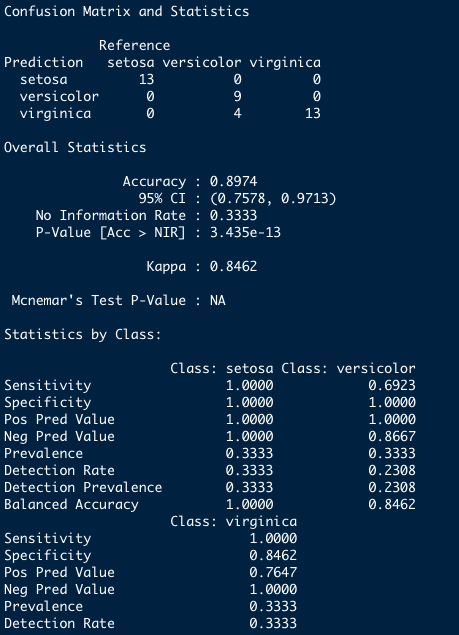
Matriz de confusão
Função confusao_lab() do pacote NIRtools1.
par(mar = c(1, 5, 5, 1))table(teste$Species, predicao_plsfit_exemplo_loocv) %>% NIRtools::confusao_lab(.)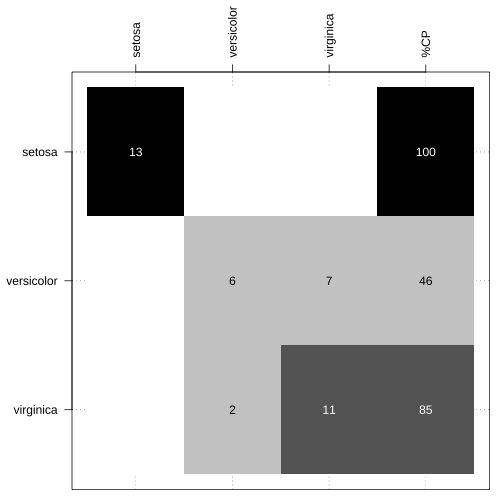
- Perdiz (2021) https://github.com/ricoperdiz/NIRtools
Resumo de construção e avaliação de modelos com a interface caret
trainControl()etrain()- controla argumentos e treina modelosgetTrainPerf()avalia o modelo com dadostreinopredict()para predizer categoria das amostras emtesteconfusionMatrix()para obter métricas de performance e matriz de confusãoconfusao_labpara plotar uma matriz de confusão no estilo LABOTAM.
Grato!
Info do sistema
## R version 4.1.2 (2021-11-01)## Platform: x86_64-apple-darwin17.0 (64-bit)## Running under: macOS Catalina 10.15.7## ## Matrix products: default## BLAS: /System/Library/Frameworks/Accelerate.framework/Versions/A/Frameworks/vecLib.framework/Versions/A/libBLAS.dylib## LAPACK: /Library/Frameworks/R.framework/Versions/4.1/Resources/lib/libRlapack.dylib## ## locale:## [1] en_US.UTF-8/en_US.UTF-8/en_US.UTF-8/C/en_US.UTF-8/en_US.UTF-8## ## attached base packages:## [1] stats graphics grDevices utils datasets methods base ## ## other attached packages:## [1] pls_2.8-0 MASS_7.3-54 DiagrammeR_1.0.8 ## [4] metathis_1.1.1 data.table_1.14.2 magrittr_2.0.2 ## [7] caret_6.0-90 lattice_0.20-45 yardstick_0.0.9 ## [10] workflowsets_0.1.0 workflows_0.2.4 tune_0.1.6 ## [13] tidyr_1.2.0 tibble_3.1.6 rsample_0.1.1 ## [16] recipes_0.1.17 purrr_0.3.4 parsnip_0.1.7 ## [19] modeldata_0.1.1 infer_1.0.0 dplyr_1.0.7 ## [22] dials_0.1.0 scales_1.1.1 broom_0.7.12 ## [25] tidymodels_0.1.4 ggplot2_3.3.5 xaringanthemer_0.4.1## ## loaded via a namespace (and not attached):## [1] colorspace_2.0-2 ellipsis_0.3.2 class_7.3-19 ## [4] showtext_0.9-4 proxy_0.4-26 xaringanExtra_0.5.2 ## [7] rstudioapi_0.13 showtextdb_3.0 listenv_0.8.0 ## [10] furrr_0.2.3 farver_2.1.0 xaringan_0.22 ## [13] prodlim_2019.11.13 fansi_1.0.2 lubridate_1.8.0 ## [16] xml2_1.3.3 codetools_0.2-18 splines_4.1.2 ## [19] knitr_1.37 jsonlite_1.7.3 pROC_1.18.0 ## [22] icons_0.2.0 compiler_4.1.2 backports_1.4.1 ## [25] assertthat_0.2.1 Matrix_1.3-4 fastmap_1.1.0 ## [28] cli_3.1.1 visNetwork_2.1.0 NIRtools_0.0.0.9006 ## [31] htmltools_0.5.2 tools_4.1.2 gtable_0.3.0 ## [34] glue_1.6.1 reshape2_1.4.4 rappdirs_0.3.3 ## [37] Rcpp_1.0.8 jquerylib_0.1.4 DiceDesign_1.9 ## [40] vctrs_0.3.8 nlme_3.1-153 blogdown_1.7 ## [43] iterators_1.0.14 timeDate_3043.102 gower_1.0.0 ## [46] xfun_0.29 stringr_1.4.0 globals_0.14.0 ## [49] lifecycle_1.0.1 future_1.23.0 ipred_0.9-12 ## [52] parallel_4.1.2 RColorBrewer_1.1-2 yaml_2.2.2 ## [55] sass_0.4.0 rpart_4.1-15 stringi_1.7.6 ## [58] highr_0.9 foreach_1.5.2 e1071_1.7-9 ## [61] lhs_1.1.3 hardhat_0.2.0 lava_1.6.10 ## [64] rlang_1.0.1 pkgconfig_2.0.3 evaluate_0.14 ## [67] htmlwidgets_1.5.4 labeling_0.4.2 tidyselect_1.1.1 ## [70] parallelly_1.30.0 plyr_1.8.6 bookdown_0.24.9 ## [73] R6_2.5.1 generics_0.1.2 DBI_1.1.2 ## [76] pillar_1.7.0 whisker_0.4 withr_2.4.3 ## [79] survival_3.2-13 nnet_7.3-16 future.apply_1.8.1 ## [82] crayon_1.4.2 utf8_1.2.2 rmarkdown_2.11.22 ## [85] sysfonts_0.8.5 grid_4.1.2 ModelMetrics_1.2.2.2## [88] digest_0.6.29 stats4_4.1.2 GPfit_1.0-8 ## [91] munsell_0.5.0 bslib_0.3.1